Integrierte Mechanistische Modelle (Perspektivgruppe)
Pflanzenwissenschaften leisten einen wesentlichen Beitrag zum Erhalt der Sortenvielfalt sowie zur nachhaltigen und effizienten Produktion von Nahrungsmittel, Energie und Rohstoffen. Dies ist aber nur möglich, wenn wir das Erbgut von Nutzpflanzen wie Weizen, Gerste und Raps entschlüsseln und Daten über den gesamten Lebenszyklus zusammen tragen - vom Samen bis zur erntereifen Pflanze.
Diese Daten helfen uns zu bestimmen, wofür jedes einzelne Gen im Erbgut verantwortlich ist und wie Gene interagieren. Dieses Wissen ermöglicht es uns zu verstehen, wie sich Veränderungen in der Umwelt und Klima oder im Erbgut auf die Merkmale einer Pflanze auswirken. Dazu gehören Ertrag, Widerstandsfähigkeit gegenüber Schädlingen, aber auch gegenüber Hitze und Trockenheit.
Datenwissenschaften und computergestützte Werkzeuge sind unverzichtbar Schlüsseltechnologien, um Informationen aus den Unmengen an Daten zu gewinnen. Nur durch deren Einsatz wird es möglich sein, Innovationen in der Pflanzenzüchtung als Antwort auf drängende Fragen zur Welternährung sowie zur Rohstoff- und Energieversorgung zu entwickeln.
Die Perspektivgruppe IMM nutzt mathematische Modelle und computergestützter Biologie zur Entschlüsselung molekularer Mechanismen, die an Wachstums- und Entwicklungsprozessen der Pflanze beteiligt sind und den Stoffwechsel regulieren. Aufgrund der Komplexität dieser Prozesse und der Vielzahl und Vielfalt von Daten sind integrative Ansätze notwendig, die Daten in ihrem Zusammenhang beschreiben und untersuchen. Im Zentrum dieser integrativen Ansätze stehen sogenannte Stoffwechsel-Netzwerke. Der Stoffwechsel spiegelt die Ausprägung der Erbinformationen wider und beeinflusst auf direkten Weg die Merkmale einer Pflanze. Neben Methoden zur Analyse metabolischer Modelle, kommen eine Reihe Rechner-basierter und mathematischer Werkzeuge zum Einsatz. Dazu gehören Graphentheorie, statistische Verfahren, maschinelles Lernen, Konzepte aus der Systemtheorie und Regelungstechnik, sowie stochastische und deterministische Modellierungsansätze.
Ziel des integrativen Ansatzes ist es nicht nur Wachstums- und Entwicklungsprozesse zu entschlüsseln und zu verstehen, sondern auch Modelle zu entwickeln die es ermöglichen die Leistungsfähigkeit einer Pflanze in verschieden Umwelten vorherzusagen. Diese Daten- und Mechanismen-basierte Vorhersagemodelle sind in der Pflanzenzüchtung essentiell um möglichst schnell und effizient auf neue klimatische Bedingungen im Zuge des Klimawandels zu reagieren.
scroll top
Projekte
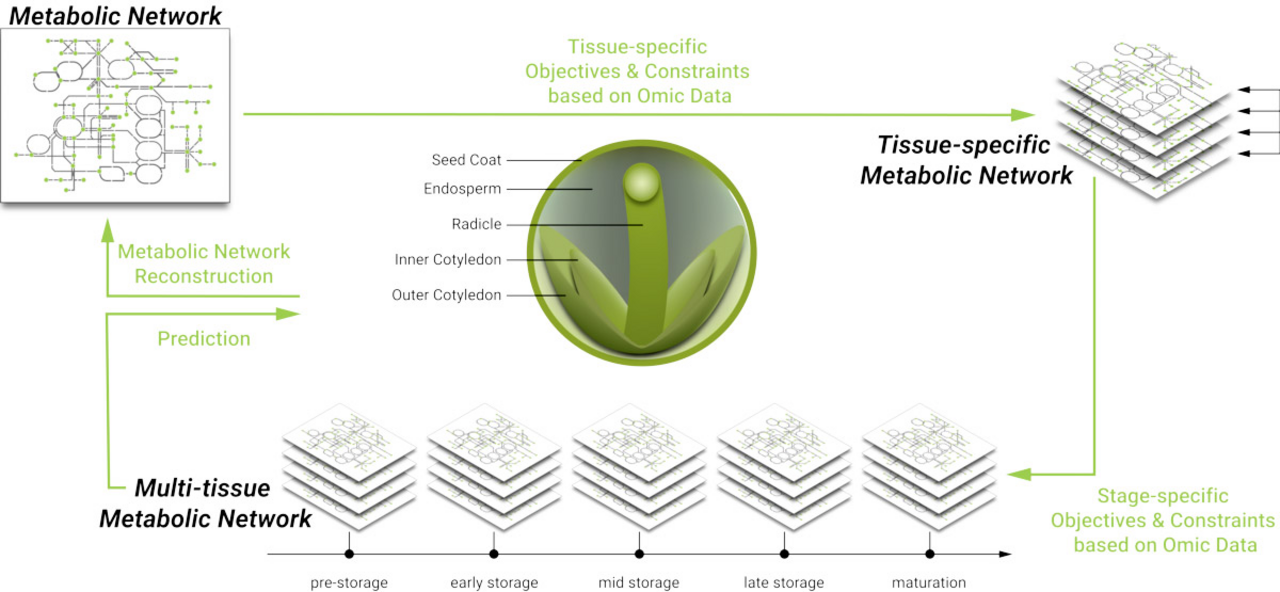
Samenentwicklung verstehen durch die Anwendung Metabolischer Modelle als Integrationswerkzeug
Die Perspektivgruppe IMM ist Teil des vom BMBF geförderten AVATARS-Konsortiums. Dieses hat das Ziel, die Entwicklung von Rapssamen und gesammelte Daten in einer VR/AR Umgebung erlebbar zu machen. Dazu werden zunächst zahlreiche Versuchsreihen auf dem Feld und Gewächshaus durchgeführt in Kombination mit umfangreichen molekularen Analysen des Rapssaatgutes in verschiedenen Entwicklungsstadien. Der Fokus von IMM liegt darauf, die gewonnenen Daten umfassend zu analysieren.
Im Zentrum des Ansatzes steht dabei ein Raps-Stoffwechselmodell. Konkret geht es darum, Daten aus einzelnen molekularen Ebenen zu verknüpfen. Veränderungen auf jeder dieser Ebenen müssen durch Veränderungen im Stoffwechsel verarbeitet werden. Der Ansatz zielt in erster Linie darauf ab, besser zu verstehen, wie metabolische Veränderungen die Samenentwicklung antreiben. Schließlich soll ein Modell entwickelt werden, das es Züchtern ermöglicht, die Keimfähigkeit unter verschiedenen klimatischen Bedingungen vorherzusagen.
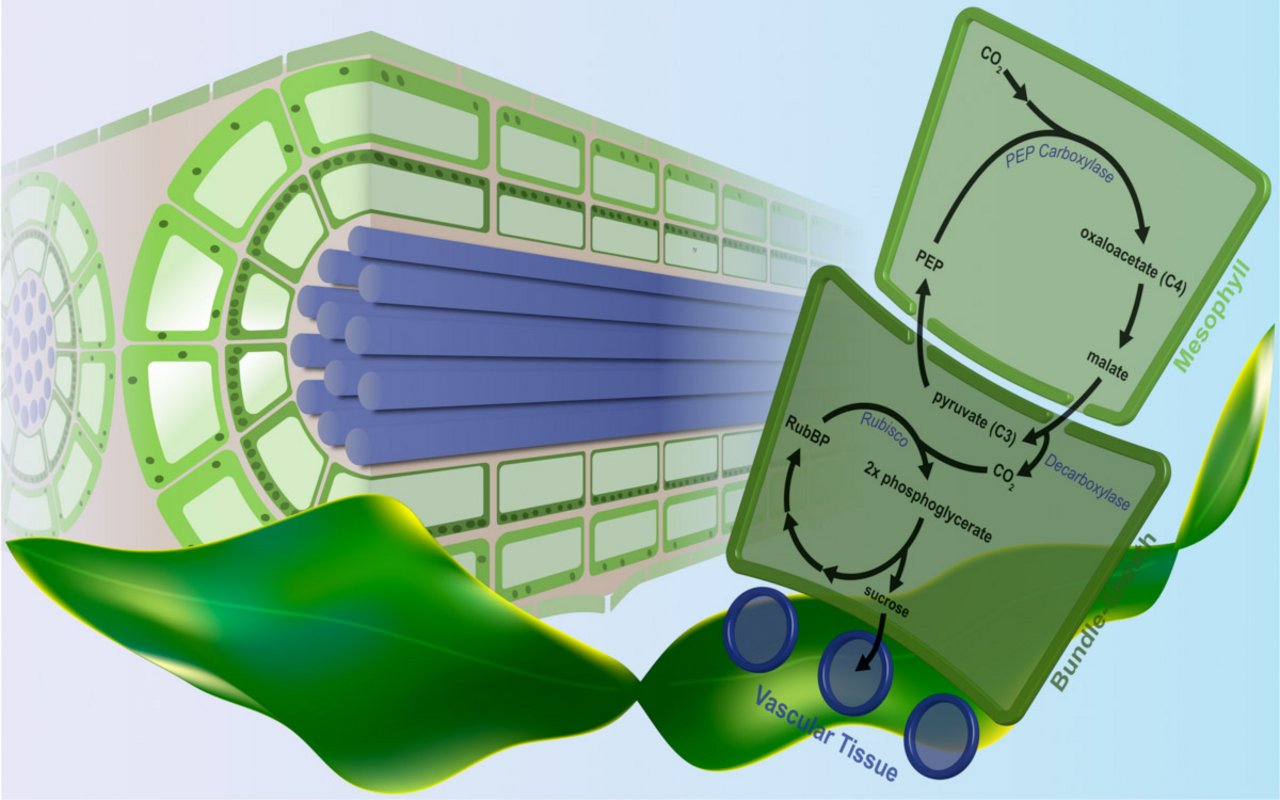
Ein Blick in die Evolution der Photosynthese mittels metabolischer Netzwerke
Alle Pflanzen, Algen und auch einige Bakterien betreiben Photosynthese, jedoch läuft der Photosynthese-Prozess nicht überall gleich ab. Die am weitesten verbreitete Form der Photosynthese in Pflanzen ist die C3-Photosynthese. Trotz der Dominanz der C3-Photosynthese nutzt diese nicht die effizienteste Form der Kohlenstofffixierung.
Eine Alternative, die C4-Photosynthese, wurde unabhängig voneinander mindestens 62-mal in 19 verschiedenen Pflanzenfamilien entwickelt. Pflanzen mit der C4-Eigenschaft verstärken ihre Kohlenstofffixierung durch eine biochemische Pumpe. Als Ergebnis haben C4-Pflanzen, zum Beispiel Mais, erhöhte Wachstumsraten. Stoffwechselnetzwerke kamen zum Einsatz, um die beiden Arten der Photosynthese zu modellieren. Deren Simulationen bildet den evolutionären Verlauf von der C3- zur C4-Photosynthese in Abhängigkeit vom Kohlenstoffdioxid-Gehalt ab.
Das Modell prognostizierte außerdem eine Art Zwischenstand als optimale Lösung unter bestimmten Voraussetzungen und erklärte, warum so viele verschiedene Varianten der C4-Photosynthese existieren. Es zeigte außerdem, dass Stickstoff und Licht spezielle Parameter sind, die eine Rolle in der Entwicklung der C4-Photosynthese spielten. Die Studie zeigt, wie wirkungsvoll die Anwendung von Stoffwechselmodellen für die Erforschung der Evolution komplexer Eigenschaften in Pflanzen ist. Gleichzeitig ebnet die erfolgreiche Analyse des C4-Entwicklungsprozesses den Weg für eine detaillierte Untersuchung der C4-Evolution und des C4-Stoffwechsels, und beleuchtet neue Ziele für zukünftige Zuchtbemühungen bei C4-Nutzpflanzen.
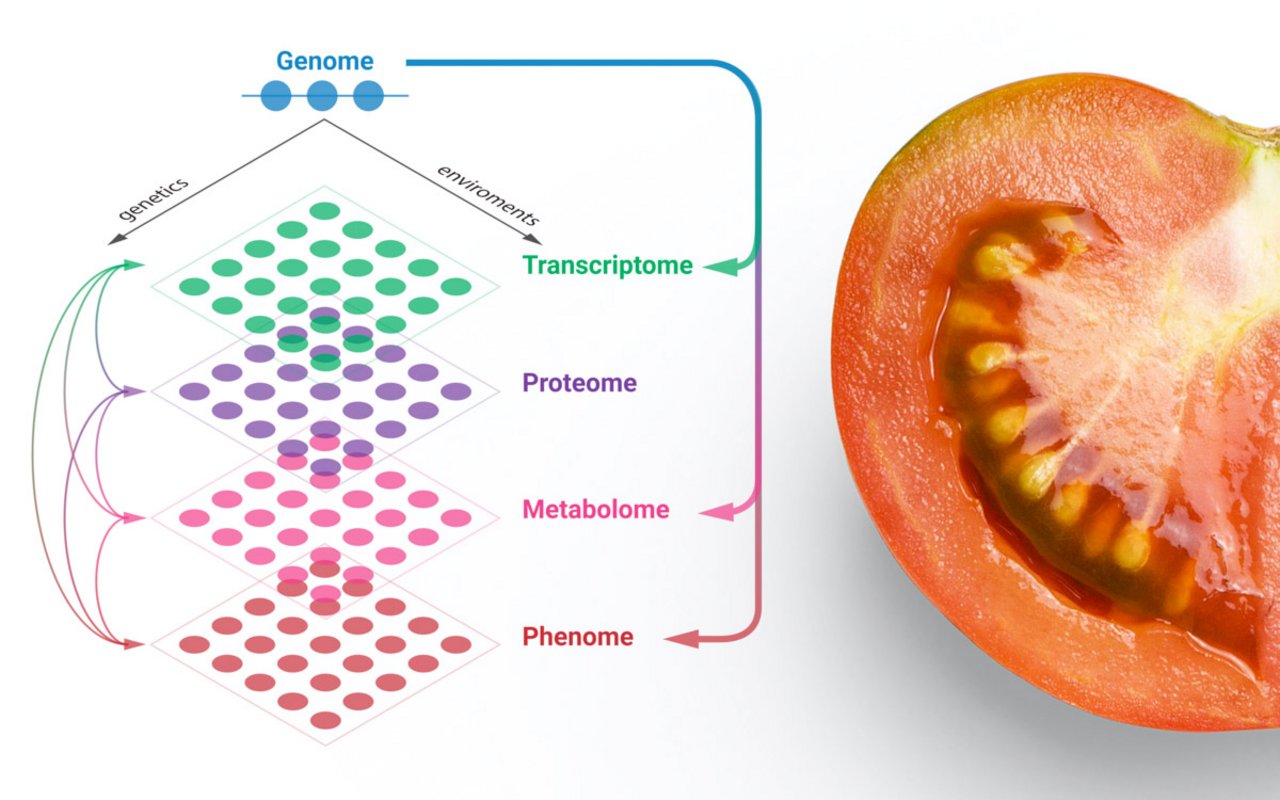
Entschlüsselung der Regulationsmechanismen bei Fruchtentwicklung in Tomaten
Die Fruchtentwicklung untergliedert sich im Wesentlichen in zwei Phasen: Wachstum und Reifung. Während des Wachstums nimmt die Fruchtmasse stetig zu bis die Frucht ihre finale Form und Größe erreicht hat. Der Reifungsprozess beginnt mit der orange-rötlichen Färbung der Frucht. Während der Reifung findet ein Umbau der Inhaltsstoffe in der Frucht statt, welche die Tomate erst genießbar und so beliebt machen.
Voraussetzung für die Fruchtentwicklung sind komplexe molekulare Veränderungen, die durch streng getaktete regulatorischen Mechanismen kontrolliert werden. Einige Komponenten des regulatorischen Netzwerkes sind bekannt, in dessen Zentrum das Pflanzenhormon Ethylen steht,. Daten zu verschiedenen Entwicklungsstadien zeigen aber eine Reihe weiterer Veränderungen, dessen Regulationsmechanismen bisher unbekannt sind.
Ziel dieses Projektes ist es verschiedene Datensätze zu integrieren, um bisher unbekannte Regulationsmechanismen zu entschlüsseln. Zu diesem Zweck kombinieren wir statistischen Datenanalyse mit Prozess-basierten mathematischen Modellen. Dieser Workflow ermöglicht es vorherzusagen, wie sich Veränderungen im Erbgut auf die einzelnen molekularen Ebenen und damit den Wachstums- und Reifungsprozess auswirken. Diese Information sind auch für den Züchter wertvoll, deren Interesse darin besteht u.a. Nährstoffgehalt, Geschmack, Ertrag und Haltbarkeit zu optimieren.
scroll top
Mitarbeitende
scroll top
Publikationen
Blätke M-A, Beier S, Scholz U, Gladilin E, Szymanski J J (Eds.):
Front. Plant Sci., Frontiers Research Topic “Advances in Applied Bioinformatics in Crops." Lausanne: Frontiers Media SA (2021) https://dx.doi.org/10.3389/978-2-88966-620-1
Blätke M-A, Szymanski J J, Gladilin E, Scholz U, Beier S:
Editorial: Advances in applied bioinformatics in crops. Front. Plant Sci. 12 (2021) 640394. https://dx.doi.org/10.3389/fpls.2021.640394
Sahu A, Blätke M-A, Szymański J J, Töpfer N:
Advances in flux balance analysis by integrating machine learning and mechanism-based models. Comput. Struct. Biotechnol. J. 19 (2021) 4626-4640. https://doi.org/10.1016/j.csbj.2021.08.004
scroll top