In silico Genbank-Proteomik
"Alles, was Lebewesen tun, lässt sich durch das Schütteln und Rütteln von Atomen erklären."
(Richard Feynman)
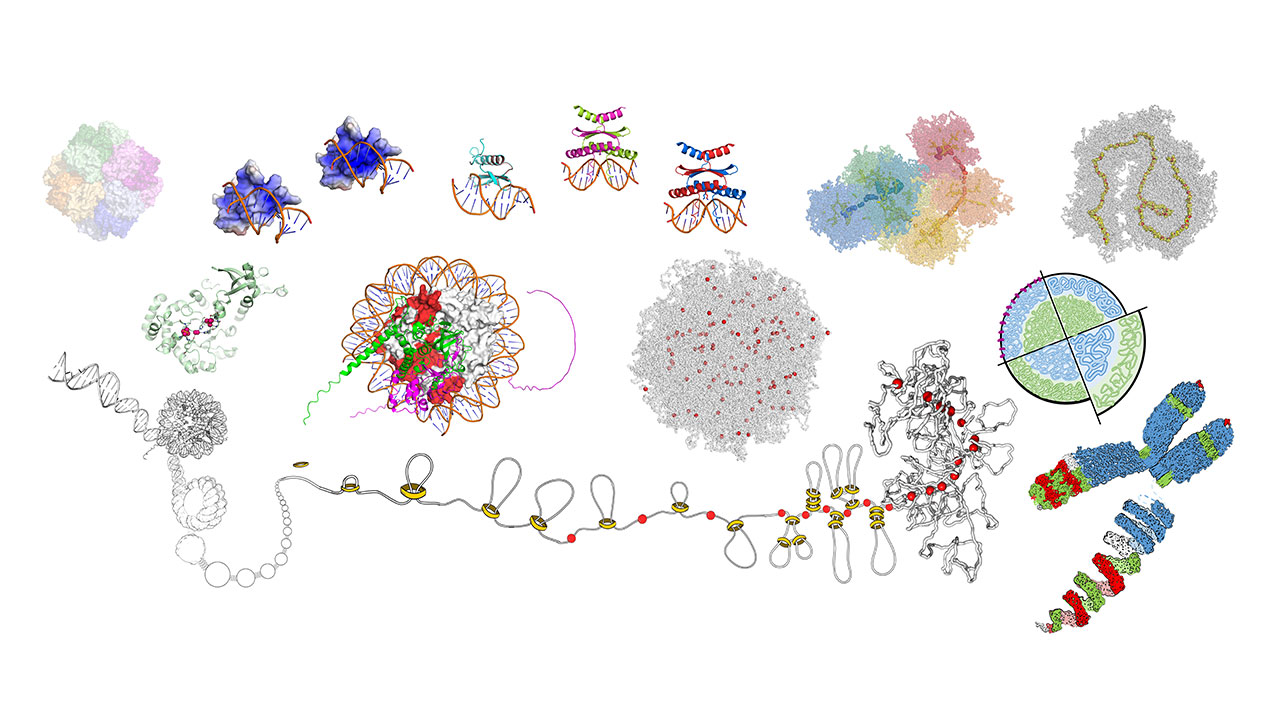
Proteine und Nukleinsäuren sind für die molekularen Prozesse des Lebens unerlässlich. Sie sind beide polymere Biomoleküle, deren Makroanordnungen Funktionen verleihen, die über die ihrer kleinsten Teile, der Aminosäuren bzw. Nukleotide, hinausgehen.
Mit den aktuellen Entwicklungen in der Genomik werden viele Informationen über die Sequenzen von Aminosäuren und Nukleotiden gewonnen. Die Aufgabe unserer Gruppe besteht darin, diese Informationen in Strukturmodelle von Proteinen und Chromosomen zu übersetzen. Solche Modelle sind wichtig, um zu verstehen, wie diese Makromoleküle funktionieren und interagieren. Unser Ziel ist es, gemeinsame und variable molekulare Merkmale und Mechanismen in verschiedenen Pflanzenarten zu finden, um zu klären, wie sich Proteine und Chromosomen strukturell entwickeln und an ihre sich verändernde Umgebung anpassen.
Die Gruppe trägt zum IPK-Forschungsthema „Genomvielfalt und Evolution“ bei.
scroll top
Projekte
In Silico Genbank Proteomik
Proteine sind die Arbeitskräfte der Zellen. Sie sind das Bindeglied zwischen der im Genom bewahrten Information und den verschiedenen Phänotypen, die wir beobachten. Mit den jüngsten Fortschritten der künstlichen Intelligenz sind wir nun in der Lage, Strukturmodelle für alle Proteine einer Art zu erstellen: ihr Proteom. Unser Ziel ist es, die strukturelle Variabilität in verschiedenen Proteomen zu charakterisieren, indem wir sequenzierte Genome mit den annotierten Phänotypen vergleichen. Dazu nutzen wir die jüngste Entwicklung von AlphaFold und die große Anzahl an Akzessionen von Nutzpflanzen in der Ex-situ-Genbank des IPK. Wir konzentrieren uns zunächst auf Gerste und arbeiten daran, die strukturellen Merkmale von Proteinen zu identifizieren, die sie von anderen Arten unterscheiden, und inwieweit sie sich bei Gerstenvarianten ändern. Ein Ergebnisssolcher explorativen Analysen ist der Aufbau einer Datenbank mit strukturellen Annotationen und Vorhersagen für jedes Protein. Hauptzweck ist es, vergleichende Analysen zwischen verschiedenen Proteinen zu ermöglichen und die Auswirkungen von Mutationen vorherzusagen. In Zusammenarbeit mit anderen Gruppen des IPK können unsere Modelle und die daraus gezogenen Schlüsse experimentell überprüft werden. Beispiele sind Transkriptionsfaktoren und Proteine des Kinetochor-Komplexes.
Literatur:
journals.plos.org/plosone/article?id=10.1371/journal.pone.0192826
Polymermodelle für die Dynamik von Chromosomen
Im dreidimensionalen Raum sehen lange DNA-Moleküle wie Wollknäule aus. Die Art und Weise, wie sie verdichtet werden, ändert sich während des Zellzyklus und variiert zwischen Zelltypen und Arten. Diese variable Organisation erleichtert die Genregulation und die Zellteilung. In Zusammenarbeit mit Experten für die Konformationserfassung von Chromosomen und für Mikroskopie, können wir die räumliche Organisation von Chromosomen mit ungeahnten Details darstellen. Ziel dieses Projekts ist es, polymere Modelle von Chromosomen zu erstellen, die die Mechanismen der Konformationsänderungen entschlüsseln. Pflanzen sind ideal für die Konformationserfassung von Chromosomen, da diese sich zwischen Arten wesentlich in der Genomgröße, Chromosomenzahl und Anteil von repetitiven Sequenzen unterscheiden können.
Literatur:
scroll top
Mitarbeitende
scroll top
Publikationen
Šimková H, Câmara A S, Mascher M:
Hi-C techniques: from genome assemblies to transcription regulation. J. Exp. Bot. (2024) Epub ahead of print. https://dx.doi.org/10.1093/jxb/erae085
scroll top