Automatisierte Pflanzenphänotypisierung
Zu unseren zentralen Aufgaben zählt das Management und die Koordination der Pflanzenphänotypisierungsinfrastruktur am IPK, in Zusammenarbeit mit internen und externen Partnern tragen wir daher zur Aufklärung des Pflanzenwachstums und seiner Plastizität unter vielfältigen Umweltbedingungen bei. Wir arbeiten eng mit der Forschungsgruppe Bioinformatik und Informationstechnologie (BIT) zusammen an der Implementierung eines Datenmanagementsystems zur standardisierten Dokumentation, nachhaltigen Speicherung und Verbreitung von Phänomik-Datensätzen nach FAIR-Prinzipien.
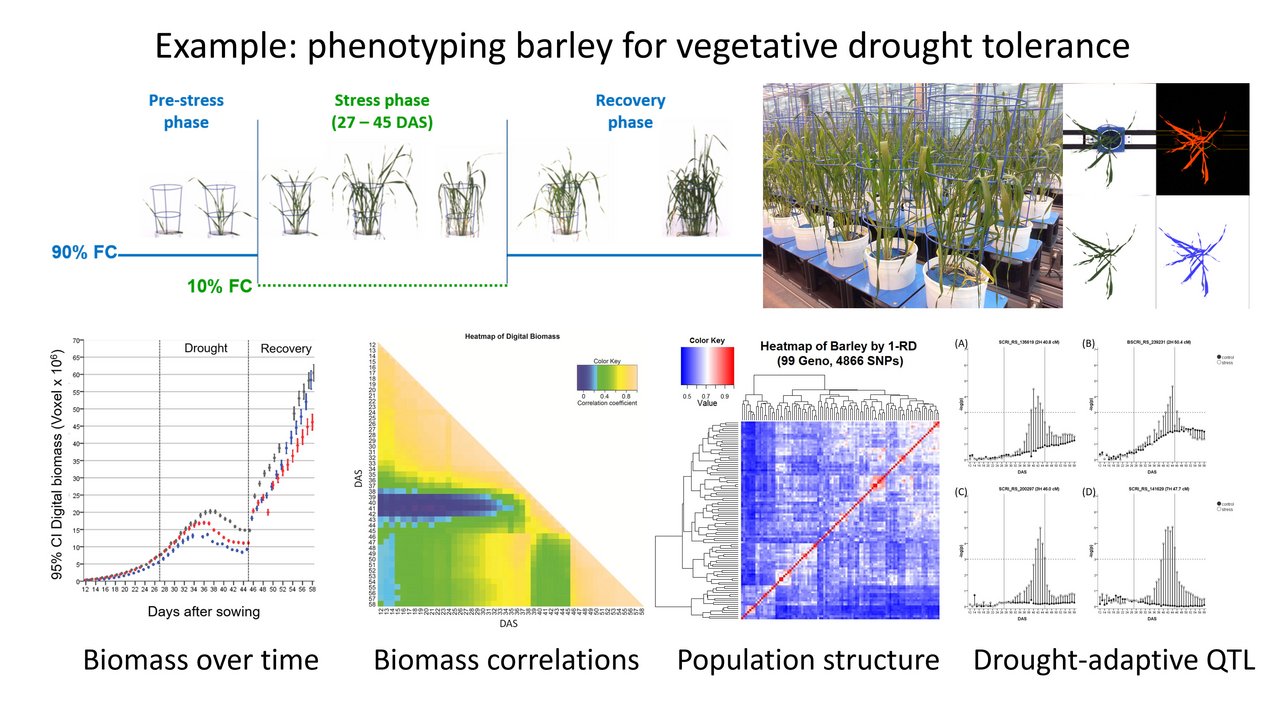
Der Fokus der eigenen Forschung liegt dabei vorwiegend auf der Erforschung der Pflanzenreaktion auf abiotischen Stress, insbesondere Trockenheit und Hitze. Diese Reaktion ist sehr umweltabhängig und erfolgt dynamisch, weshalb wir Präzisions-Phänotypisierungssysteme einsetzen, welche eine nicht-invasive Erfassung unter kontrollierten Umweltbedingungen ermöglichen. Der Schwerpunkt liegt dabei auf der Genetik und der Physiologie als auch auf der Modellierung des Pflanzenwachstums sowie der Wassernutzung (Abb. 1). Genutzte Pflanzenmaterialien sind vor allem pflanzengenetische Ressourcen von Getreide und Leguminosen, welche eine wichtige Quelle darstellen, um über züchterische Ansätze die Stresstoleranz der modernen Sorten zu verbessern für die nötige Anpassung an den fortschreitenden Klimawandel.
scroll top
Projekte
Im Rahmen des Deutschen Pflanzenphänotypisierungs Netzwerks (DPPN) erweitern wir die vorhandene Infrastruktur zur Pflanzenphänotypisierung und stellen diese Nutzern aus Forschung und Industrie im Rahmen von Kollaborationsprojekten und Transnational Access Projekten (EPPN2020) zur Verfügung.
STARGATE
Im Rahmen von STARGATE werden wir unseren Partner in Portugal (UCP) dabei unterstützen, ein etabliertes Exzellenzzentrum für den Einsatz von Sensoren, Multi-omics und Pflanzenphänotypisierungstechnologien zu werden.
BRACE
In BRACE untersuchen wir Mechanismen der Nachhaltigkeit unter abiotischem Stress in der ersten Wildgersten-Multi-Eltern-Nested-Association-Mapping (NAM)-Population (HEB-25) unter Verwendung moderner automatisierter Phänotypisierungsansätze.
BOLERO
Die Wurzelentwicklung von Kaffee und Kakaopflanzen steht im Fokus von BOLERO, wo wir mit Hilfe der automatischen Phänotypisierung die Dynamik der Wurzelentwicklung untersuchen sowie die Reaktion auf ein unterschiedliches Nährstoffangebot.
INCREASE
Im Rahmen von INCREASE tragen wir zur Förderung der landwirtschaftlichen Biodiversität in Europa bei, indem wir Wachstum und Trockentoleranz von 200 genetischen Ressourcen der Kichererbse mittels Präzisionsphänotypisierung evaluieren und ein europaweites Citizen Science Experiment mit 1.000 genetischen Ressourcen der Ackerbohne durchführen. Seit drei Jahren sind Bürgerinnen und Bürger in ganz Europa aufgerufen, sich am Experiment zur Bohnenvielfalt zu beteiligen. Das IPK lud dafür am 21. Juli 2023 100 deutsche Teilnehmerinnen und Teilnehmer zu einem Bürgertag an das Institut ein.
Citizen Science Day 2023
scroll top
Mitarbeitende
scroll top
Publikationen
Bienert M D, Junker A, Melzer M, Altmann T, von Wirén N, Bienert G P:
Boron deficiency responses in maize (Zea mays L.) roots. J. Plant Nutr. Soil Sci. (2026) Epub ahead of print. https://doi.org/10.1002/jpln.202300173
Laura T, Rosaria T M, Silvia R, Tea S, Teresa A M, Lorenzo B, Luciana G, Salar S, Laura R, Leonardo R G:
Construction of a living library of Solanum tomentosum introgression lines into the S. melongena genome: a tool to exploit novel genetic diversity for eggplant breeding. Hortic. Plant J. (2026) Epub ahead of print. https://doi.org/10.1016/j.hpj.2025.04.019
Narisetti N, Neumann K, Altmann T, Stolzenburg F, von Wirén N, Giehl R F, Gladilin E:
A multi-class deep learning segmentation approach for automated analysis of axial and lateral roots in barley plants. Comput. Electron. Agric. 240 (2026) 111157. https://dx.doi.org/10.1016/j.compag.2025.111157
Alcock T D, Bienert M D, Junker A, Meyer R C, Tschiersch H, Kudamala S, von Wirén N, Altmann T, Bienert G P:
Arabidopsis thaliana exhibits wide within-species variation in tolerance to boron limitation and root and shoot trait resilience associate with a pleiotropic locus. New Phytol. 248 (2025) 2444-2465. https://doi.org/10.1111/nph.70570
Fernández-Alonso M J, de Celis M, Belda I, Palomino J, García C, Gaitán J, Wang J-T, Abdala-Roberts L, Alfaro F D, Angulo-Pérez D F, Arthikala M-K, Chalasani D, Corwin J, Gui-Lan D, Hernandez-Lopez A, Nanjareddy K, Nayaka S C, Pasari B, Patro T S S K, Podile A R, Quijano-Medina T, Rivera D S, Sarma P V S R N, Shaaf S, Trivedi P, Yang Q, Yin Y, Zaady E, Zhu Y-G, Singh B K, Delgado-Baquerizo M, García-Palacios P, Milla R:
Native edaphoclimatic regions shape soil communities of crop wild progenitors. ISME Commun. 5 (2025) ycaf143. https://dx.doi.org/10.1093/ismeco/ycaf143
Gladilin E, Narisetti N, Neumann K, Altmann T:
End-to-end deep learning approach to automated phenotyping of greenhouse-grown plant shoots. Agronomy 15 (2025) 1117. https://dx.doi.org/10.3390/agronomy15051117
Inoue M, Situmorang A, Kelly J H, Chen W, Zhou H, Ferrario C C, Gregis V, Vajani A, Shaaf S, Biswas A, Alqusumi R, Waters M T, Tucker M R, Zhang D, Watts-Williams S J, Janiak A, Marzec M, Chmielewska B, Rossini L, Yoneyama K, Brewer P B:
LATERAL BRANCHING OXIDOREDUCTASE specificity for strigolactone branching inhibition in barley. J. Exp. Bot. 76 (2025) 5367-5381. https://dx.doi.org/10.1093/jxb/eraf285
Lauterberg M M:
Utilizing high-throughput phenotyping to dissect plant genetic resources of wheat and chickpea under drought stress. (PhD Thesis) Halle/S., Martin-Luther-Universität Halle-Wittenberg, Naturwissenschaftliche Fakultät III Agrar- und Ernährungswissenschaften, Geowissenschaften und Informatik (2025) 101 pp.
Montardit-Tarda F, Casas A M, Thomas W T B, Schnaithmann F, Sharma R, Shaaf S, Campoli C, Russell J, Ramsay L, Bayer M M, Delbono S, Jääskeläinen M, Paul M, Stoddard F L, Visioni A, Flavell A J, Pillen K, Kilian B, Graner A, Rossini L, Waugh R, Cattivelli L, Schulman A H, Tondelli A, Igartua E:
New loci and candidate genes in spring two-rowed barley detected through meta-analysis of a field trial European network. Theor. Appl. Genet. 138 (2025) 158. https://dx.doi.org/10.1007/s00122-025-04934-8
Sahu A, Psaroudakis D, Rolletschek H, Neumann K, Borisjuk L, Himmelbach A, Pinninti K, Knoch D, Töpfer N, Szymanski J:
panomiX: Investigating mechanisms of trait emergence through multi-omics data integration. Plant Phenomics 7 (2025) 100131. https://doi.org/10.1016/j.plaphe.2025.100131
Schüler D, Lange M, Altmann T, Cuacos M, Arend D, D’Auria J C, Fiebig A, Kumlehn J, Neumann K, Melzer M, Rey-Mazón E, Rolletschek H, Scholz U, Willner E, Reif J C:
Data management in balance – a decade of balancing pragmatism, sustainability and innovation at plant research center IPK Gatersleben. J. Integr. Bioinform. 22 (2025) 20250012. https://dx.doi.org/10.1515/jib-2025-0012
Sharma R, Shaaf S, Neumann K, Civan P, Guo Y, Mascher M, David M, Al-Yassin A, Özkan H, Blake T, Hübner S, Castañeda-Álvarez N P, Grando S, Ceccarelli S, Baum M, Graner A, Coupland G, Pillen K, Weiss E, Mackay I J, Powell W, Kilian B:
On the origin of the late-flowering ppd-H1 allele in barley. Theor. Appl. Genet. 138 (2025) 246. https://dx.doi.org/10.1007/s00122-025-04981-1
Shi R, López-Malvar A, Knoch D, Tschiersch H, Heuermann M C, Shaaf S, Madur D, Santiago R, Balconi C, Frascaroli E, Erdal S, Palaffre C, Charcosset A, Revilla P, Altmann T:
A high-throughput phenotyping dataset for GWAS analysis of maize under combined drought and heat stress. Data Brief 62 (2025) 111947. https://dx.doi.org/10.1016/j.dib.2025.111947
Shi R, López-Malvar A, Knoch D, Tschiersch H, Heuermann M C, Shaaf S, Madur D, Santiago R, Balconi C, Frascaroli E, Erdal S, Palaffre C, Charcosset A, Revilla P, Altmann T:
Integrating high-throughput phenotyping and genome-wide association analyses to unravel Mediterranean maize resilience to combined drought and high temperatures. Plant Stress 17 (2025) 100954. https://doi.org/10.1016/j.stress.2025.100954
Soleimani S, Wichmann P, Pillen K, Neumann K, Maurer A:
Tillering plasticity of drought-stressed barley genotypes under different re-watering regimes. BMC Plant Biol. 25 (2025) 1368. https://dx.doi.org//10.1186/s12870-025-07504-8
Turek S, Skarzyńska-Łyżwa A, Aparna A, Pląder W, Riewe D, Junker A, Altmann T, Pawełkowicz M:
Multi-omics integration of transcriptome, miRNA, and metabolome uncovers molecular mechanisms of male flower development in cucumber line B10 (Cucumis sativus L.). Sci. Rep. 15 (2025) 45734. https://dx.doi.org/10.1038/s41598-025-28485-6
Ullah S, Narisetti N, Neumann K, Altmann T, Hejatko J, Gladilin E:
Automated generation of ground truth images of greenhouse-grown plant shoots using a GAN approach. Plant Methods 21 (2025) 126. https://dx.doi.org/10.1186/s13007-025-01441-1
Wonneberger R, DAuria J C, Neumann K, Hansen P B, Dieseth J A, Nielsen L K, Niemelä T, Odilbekov F, Novakazi F, Bengtsson T, CResWheat Consortium:
Integrating metabolomics and high-throughput phenotyping to elucidate metabolic and phenotypic responses to early-season drought stress in Nordic spring wheat. BMC Plant Biol. 25 (2025) 987. https://dx.doi.org/10.1186/s12870-025-06914-y
Cortinovis G, Vincenzi L, Anderson R, Marturano G, Marsh J I, Bayer P E, Rocchetti L, Frascarelli G, Lanzavecchia G, Pieri A, Benazzo A, Bellucci E, Di Vittori V, Nanni L, Ferreira Fernández J J, Rossato M, Aguilar O M, Morrell P L, Rodriguez M, Gioia T, Neumann K, Alvarez Diaz J C, Gratias A, Klopp C, Bitocchi E, Geffroy V, Delledonne M, Edwards D, Papa R:
Adaptive gene loss in the common bean pan-genome during range expansion and domestication. Nat. Commun. 15 (2024) 6698. https://dx.doi.org/10.1038/s41467-024-51032-2
de Celis M, Fernandéz-Alonso M J, Belda I, García C, Ochoa-Hueso R, Palomino J, Singh B K, Yin Y, Wang J-T, Abdala-Roberts L, Alfaro F D, Angulo-Pérez D, Arthikala M-K, Corwin J, Gui-Lan D, Hernandez-Lopez A, Nanjareddy K, Pasari B, Quijano-Medina T, Rivera D S, Shaaf S, Trivedi P, Yang Q, Zaady E, Zhu Y-G, Delgado-Baquerizo M, Milla R, García-Palacios P:
The abundant fraction of soil microbiomes regulates the rhizosphere function in crop wild progenitors. Ecol. Lett. 27 (2024) e14462. https://dx.doi.org/10.1111/ele.14462
Heuermann M C, Meyer R C, Knoch D, Tschiersch H, Altmann T:
Strong prevalence of light regime-specific QTL in Arabidopsis detected using automated high-throughput phenotyping in fluctuating or constant light. Physiol. Plant. 176 (2024) e14255. https://dx.doi.org/10.1111/ppl.14255
Lauterberg M, Tschiersch H, Zhao Y, Kuhlmann M, Mücke I, Papa R, Bitocchi E, Neumann K:
Implementation of theoretical non-photochemical quenching (NPQ((T))) to investigate NPQ of chickpea under drought stress with High-throughput Phenotyping. Sci. Rep. 14 (2024) 13970. https://dx.doi.org/10.1038/s41598-024-63372-6
Pinninti K:
Transcriptomic analysis of thermomorphogenesis in tomato seedlings. (Master Thesis) Kiel, Christian-Albrechts-Universität zu Kiel (2024)
Ullah S, Panzarová K, Trtílek M, Lexa M, Máčala V, Neumann K, Altmann T, Hejátko J, Pernisová M, Gladilin E:
High-throughput spike detection in greenhouse cultivated grain crops with attention mechanisms-based deep learning models. Plant Phenomics 6 (2024) 0155. https://dx.doi.org/10.34133/plantphenomics.0155
Bellucci E, Benazzo A, Xu C, Bitocchi E, Rodriguez M, Alseekh S, Di Vittori V, Gioia T, Neumann K, Cortinovis G, Frascarelli G, Murube E, Trucchi E, Nanni L, Ariani A, Logozzo G, Shin J H, Liu C, Jiang L, Ferreira J J, Campa A, Attene G, Morrell P L, Bertorelle G, Graner A, Gepts P, Fernie A R, Jackson S A, Papa R:
Selection and adaptive introgression guided the complex evolutionary history of the European common bean. Nat. Commun. 14 (2023) 1908. https://dx.doi.org/10.1038/s41467-023-37332-z
Heuermann M C, Knoch D, Junker A, Altmann T:
Natural plant growth and development achieved in the IPK PhenoSphere by dynamic environment simulation. Nat. Commun. 14 (2023) 5783. https://dx.doi.org/10.1038/s41467-023-41332-4
Khodaeiaminjan M, Knoch D, Ndella Thiaw M R, Marchetti C F, Kořínková N, Techer A, Nguyen T D, Chu J, Bertholomey V, Doridant I, Gantet P, Graner A, Neumann K, Bergougnoux V:
Genome-wide association study in two-row spring barley landraces identifies QTL associated with plantlets root system architecture traits in well-watered and osmotic stress conditions. Front. Plant Sci. 14 (2023) 1125672. https://dx.doi.org/10.3389/fpls.2023.1125672
Lauterberg M, Tschiersch H, Papa R, Bitocchi E, Neumann K:
Engaging precision phenotyping to scrutinize vegetative drought tolerance and recovery in chickpea plant genetic resources. Plants 12 (2023) 2866. https://dx.doi.org/10.3390/plants12152866
Neumann K, Schulthess A W, Bassi F M, Dhanagond S, Khlestkina E, Börner A, Graner A, Kilian B:
Genomic approaches to using diversity for the adaptation of modern varieties of wheat and barley to climate change. In: Ghamkhar K, Williams W, Brown A H D (Eds.): Plant Genetic Resources for the 21st Century. The OMICS Era. New York: Apple Academic Press (2023) ISBN 9781774910825, 47-78. https://dx.doi.org/10.1201/9781003302957
Papalini S, Di Vittori V, Pieri A, Allegrezza M, Frascarelli G, Nanni L, Bitocchi E, Bellucci E, Gioia T, Pereira L G, Susek K, Tenaillon M, Neumann K, Papa R:
Challenges and opportunities behind the use of Herbaria in paleogenomics studies. Plants 12 (2023) 3452. https://dx.doi.org/10.3390/plants12193452
Peterson A, Kishchenko O, Kuhlmann M, Tschiersch H, Fuchs J, Tikhenko N, Schubert I, Nagel M:
Cryopreservation of duckweed genetic diversity as model for long-term preservation of aquatic flowering plants. Plants 12 (2023) 3302. https://dx.doi.org/10.3390/plants12183302
Radchuk V, Belew Z M, Gündel A, Mayer S, Hilo A, Hensel G, Sharma R, Neumann K, Ortleb S, Wagner S, Muszynska A, Crocoll C, Xu D, Hoffie I, Kumlehn J, Fuchs J, Peleke F F, Szymanski J J, Rolletschek H, Nour-Eldin H H, Borisjuk L:
SWEET11b transports both sugar and cytokinin in developing barley grains. Plant Cell 35 (2023) 2186-2207. https://dx.doi.org/10.1093/plcell/koad055
Shi R, Seiler C, Knoch D, Junker A, Altmann T:
Integrated phenotyping of root and shoot growth dynamics in maize reveals specific interaction patterns in inbreds and hybrids and in response to drought. Front. Plant Sci. 14 (2023) 1233553. https://dx.doi.org/10.3389/fpls.2023.1233553
Zahn T, Zhu Z, Ritoff N, Krapf J, Junker A, Altmann T, Schmutzer T, Tüting C, Kastritis P L, Babben S, Quint M, Pillen K, Maurer A:
Novel exotic alleles of EARLY FLOWERING 3 determine plant development in barley. J. Exp. Bot. 74 (2023) 3630-3650. https://dx.doi.org/10.1093/jxb/erad127
Amitrano C, Junker A, DAgostino N, De Pascale S, De Micco V:
Integration of high-throughput phenotyping with anatomical traits of leaves to help understanding lettuce acclimation to a changing environment. Planta 256 (2022) 68. https://dx.doi.org/10.1007/s00425-022-03984-2
Arend D, Psaroudakis D, Memon J A, Rey-Mazón E, Schüler D, Szymanski J J, Scholz U, Junker A, Lange M:
From data to knowledge - big data needs stewardship, a plant phenomics perspective. Plant J. 111 (2022) 335-347. https://dx.doi.org/10.1111/tpj.15804
Badaeva E D, Konovalov F A, Knüpffer H, Fricano A, Ruban A S, Kehel Z, Zoshchuk S A, Surzhikov S A, Neumann K, Graner A, Hammer K, Filatenko A, Bogaard A, Jones G, Özkan H, Kilian B:
Genetic diversity, distribution and domestication history of the neglected GGAtAt genepool of wheat. Theor. Appl. Genet. 135 (2022) 755–776. https://dx.doi.org/10.1007/s00122-021-03912-0
Deblieck M, Szilagyi G, Andrii F, Saranga Y, Lauterberg M, Neumann K, Krugman T, Perovic D, Pillen K, Ordon F:
Dissection of a grain yield QTL from wild emmer wheat reveals sub-intervals associated with culm length and kernel number. Front. Genet. 13 (2022) 955295. https://dx.doi.org/10.3389/fgene.2022.955295
Langstroff A, Heuermann M C, Stahl A, Junker A:
Opportunities and limits of controlled-environment plant phenotyping for climate response traits. Theor. Appl. Genet. 135 (2022) 1–16. https://dx.doi.org/10.1007/s00122-021-03892-1
Lauterberg M, Saranga Y, Deblieck M, Klukas C, Krugman T, Perovic D, Ordon F, Graner A, Neumann K:
Precision phenotyping across the life cycle to validate and decipher drought-adaptive QTLs of wild emmer wheat (Triticum turgidum ssp. dicoccoides) introduced into elite wheat varieties. Front. Plant Sci. 13 (2022) 965287. https://dx.doi.org/10.3389/fpls.2022.965287
Narisetti N, Henke M, Neumann K, Stolzenburg F, Altmann T, Gladilin E:
Deep learning based greenhouse image segmentation and shoot phenotyping (DeepShoot). Front. Plant Sci. 13 (2022) 906410. https://dx.doi.org/10.3389/fpls.2022.906410
Bellucci E, Aguilar O M, Alseekh S, Bett K, Brezeanu C, Cook D, De la Rosa L, Delledonne M, Dostatny D F, Ferreira J J, Geffroy V, Ghitarrini S, Kroc M, Kumar Agrawal S, Logozzo G, Marino M, Mary-Huard T, McClean P, Meglič V, Messer T, Muel F, Nanni L, Neumann K, Servalli F, Străjeru S, Varshney R K, Vasconcelos M W, Zaccardelli M, Zavarzin A, Bitocchi E, Frontoni E, Fernie A R, Gioia T, Graner A, Guasch L, Prochnow L, Opperman M, Susek K, Tenaillon M, Papa R:
The INCREASE project: Intelligent collections of food-legume genetic resources for European agrofood systems. Plant J. 108 (2021) 646-660. https://dx.doi.org/10.1111/tpj.15472
Cortinovis G, Oppermann M, Neumann K, Graner A, Gioia T, Marsella M, Alseekh S, Fernie A R, Papa R, Bellucci E, Bitocchi E:
Towards the development, maintenance, and standardized phenotypic characterization of single-seed-descent genetic resources for common bean. Curr. Protoc. 1 (2021) e133. https://dx.doi.org/10.1002/cpz1.133
Dodig D, Božinović S, Nikolić A, Zorić M, Vančetović J, Ignjatović-Micić D, Delić N, Weigelt-Fischer K, Altmann T, Junker A:
Dynamics of maize vegetative growth and drought adaptability using image-based phenotyping under controlled conditions. Front. Plant Sci. 12 (2021) 652116. https://dx.doi.org/10.3389/fpls.2021.652116
Fadoul H E, Martínez Rivas F J, Neumann K, Balazadeh S, Fernie A R, Alseekh S:
Comparative molecular and metabolic profiling of two contrasting wheat cultivars under drought stress. Int. J. Mol. Sci. 22 (2021) 13287. https://dx.doi.org/10.3390/ijms222413287
Henke M, Neumann K, Altmann T, Gladilin E:
Semi-automated ground truth segmentation and phenotyping of plant structures using k-means clustering of eigen-colors (kmSeg). Agriculture 11 (2021) 1098. https://dx.doi.org/10.3390/agriculture11111098
Kroc M, Tomaszewska M, Czepiel K, Bitocchi E, Oppermann M, Neumann K, Guasch L, Bellucci E, Alseekh S, Graner A, Fernie A R, Papa R, Susek K:
Towards development, maintenance, and standardized phenotypic characterization of single-seed-descent genetic resources for lupins. Curr. Protoc. 1 (2021) e191. https://dx.doi.org/10.1002/cpz1.191
Li M, Hensel G, Melzer M, Junker A, Tschiersch H, Ruwe H, Arend D, Kumlehn J, Börner T, Stein N:
Mutation of the ALBOSTRIANS ohnologous gene HvCMF3 impairs chloroplast development and thylakoid architecture in barley. Front. Plant Sci. 12 (2021) 732608. https://dx.doi.org/10.3389/fpls.2021.732608
Li M, Ruwe H, Melzer M, Junker A, Hensel G, Tschiersch H, Schwenkert S, Chamas S, Schmitz-Linneweber C, Börner T, Stein N:
The Arabidopsis AAC proteins CIL and CIA2 are sub-functionalized paralogs involved in chloroplast development. Front. Plant Sci. 12 (2021) 681375. https://dx.doi.org/10.3389/fpls.2021.681375
Mayer G, Müller W, Schork K, Uszkoreit J, Weidemann A, Wittig U, Rey M, Quast C, Felden J, Glöckner F O, Lange M, Arend D, Beier S, Junker A, Scholz U, Schüler D, Kestler H A, Wibberg D, Pühler A, Twardziok S, Eils J, Eils R, Hoffmann S, Eisenacher M, Turewicz M:
Implementing FAIR data management within the German Network for Bioinformatics Infrastructure (de.NBI) exemplified by selected use cases. Brief. Bioinform. 22 (2021) bbab010. https://dx.doi.org/10.1093/bib/bbab010
Narisetti N, Henke M, Seiler C, Junker A, Ostermann J, Altmann T, Gladilin E:
Fully-automated root image analysis (faRIA). Sci. Rep. 11 (2021) 16047. https://dx.doi.org/10.1038/s41598-021-95480-y
Pommier C, Gruden K, Junker A, Coppens F, Finkers R, Hassani-Pak K, Faria D, Hancock J M, Beier S, Costa B, Miguel C, Chaves I, Davey R, Contreras-Moreira B:
ELIXIR Plant sciences 2020-2023 Roadmap [version 1; not peer reviewed]. F1000Research 10 (2021) 145. https://doi.org/10.7490/f1000research.1118482.1
Rebola-Lichtenberg J, Streit J, Schall P, Ammer C, Seidel D:
From facilitation to competition: the effect of black locust (Robinia pseudoacacia L.) on the growth performance of four poplar-hybrids (Populus spp.) in mixed short rotation coppice. New Forest. 52 (2021) 639-656. https://dx.doi.org/10.1007/s11056-020-09813-2
Sharma S, Schulthess A W, Bassi F M, Badaeva E D, Neumann K, Graner A, Özkan H, Werner P, Knüpffer H, Kilian B:
Introducing beneficial alleles from plant genetic resources into the wheat germplasm. Biology 10 (2021) 982. https://dx.doi.org/10.3390/biology10100982
Arend D, König P, Junker A, Scholz U, Lange M:
The on-premise data sharing infrastructure e!DAL: Foster FAIR data for faster data acquisition. GigaScience 9 (2020) giaa107. https://doi.org/10.1093/gigascience/giaa107
Henke M, Junker A, Neumann K, Altmann T, Gladilin E:
A two-step registration-classification approach to automated segmentation of multimodal images for high-throughput greenhouse plant phenotyping. Plant Methods 16 (2020) 95. https://dx.doi.org/10.1186/s13007-020-00637-x
Mang C:
Characterization of high light acclimation capacity in Arabidopsis mutant candidates. (Master Thesis) Mittweida, Hochschule Mittweida, Fakultät Angewandte Computer- und Biowissenschaften (2020) 81 pp.
Mikołajczak K, Ogrodowicz P, Ćwiek-Kupczyńska H, Weigelt-Fischer K, Mothukuri S R, Junker A, Altmann T, Krystkowiak K, Adamski T, Surma M, Kuczyńska A, Krajewski P:
Image phenotyping of spring barley (Hordeum vulgare L.) RIL population under drought: selection of traits and biological interpretation. Front. Plant Sci. 11 (2020) 743. https://dx.doi.org/10.3389/fpls.2020.00743
Papoutsoglou E A, Faria D, Arend D, Arnaud E, Athanasiadis I N, Chaves I, Coppens F, Cornut G, Costa B V, Ćwiek-Kupczyńska H, Droesbeke B, Finkers R, Gruden K, Junker A, King G J, Krajewski P, Lange M, Laporte M-A, Michotey C, Oppermann M, Ostler R, Poorter H, Ramı́rez-Gonzalez R, Ramšak Ž, Reif J C, Rocca-Serra P, Sansone S-A, Scholz U, Tardieu F, Uauy C, Usadel B, Visser R G F, Weise S, Kersey P J, Miguel C M, Adam-Blondon A-F, Pommier C:
Enabling reusability of plant phenomic datasets with MIAPPE 1.1. New Phytol. 227 (2020) 260-273. https://dx.doi.org/10.1111/nph.16544
Psaroudakis D:
A multi-omics perspective on Arabidopsis drought response: discovering novel regulators with a machine learning based gene-to-phene approach. (Master Thesis) Mittweida, Hochschule Mittweida, Fakultät Angewandte Computer- und Biowissenschaften (2020) 57 pp.
Psaroudakis D, Liu F, König P, Scholz U, Junker A, Lange M, Arend D:
isa4j: a scalable Java library for creating ISA-Tab metadata [version 1; peer review: 2 approved]. F1000Research 9(ELIXIR) (2020) 1388. https://doi.org/10.12688/f1000research.27188.1
Dodig D, Bozinovic S, Nikolic A, Zoric M, Vancetovic J, Ignjatovic-Micic D, Delic N, Weigelt-Fischer K, Junker A, Altmann T:
Image-derived traits related to mid-season growth performance of maize under nitrogen and water stress. Front. Plant Sci. 10 (2019) 814. https://dx.doi.org/10.3389/Fpls.2019.00814
Ghaffar M, Schüler D, König P, Arend D, Junker A, Scholz U, Lange M:
Programmatic access to FAIRified digital plant genetic resources. J. Integr. Bioinform. 16 (2019) 20190060. https://dx.doi.org/10.1515/jib-2019-0060
Henke M, Junker A, Neumann K, Altmann T, Gladilin E:
Comparison and extension of three methods for automated registration of multimodal plant images. Plant Methods 15 (2019) 44. https://dx.doi.org/10.1186/s13007-019-0426-8
Henke M, Junker A, Neumann K, Altmann T, Gladilin E:
Comparison of feature point detectors for multimodal image registration in plant phenotyping. PLoS One 14 (2019) e0221203. https://dx.doi.org/10.1371/journal.pone.0221203
Lobet G, Paez-Garcia A, Schneider H, Junker A, Atkinson J A, Tracy S:
Demystifying roots: A need for clarification and extended concepts in root phenotyping. Plant Sci. 282 (2019) 11-13. https://doi.org/10.1016/j.plantsci.2018.09.015
Narisetti N, Henke M, Seiler C, Shi R, Junker A, Altmann T, Gladilin E:
Semi-automated Root Image Analysis (saRIA). Sci. Rep. 9 (2019) 19674. https://dx.doi.org/10.1038/s41598-019-55876-3
Henke M, Junker A, Neumann K, Altmann T, Gladilin E:
Automated alignment of multi-modal plant images using integrative phase correlation approach. Front. Plant Sci. 9 (2018) 1519. https://dx.doi.org/10.3389/fpls.2018.01519
Pommerrenig B, Junker A, Abreu I, Bieber A, Fuge J, Willner E, Bienert M D, Altmann T, Bienert G P:
Identification of rapeseed (Brassica napus) cultivars with a high tolerance to boron-deficient conditions. Front. Plant Sci. 9 (2018) 1142. https://dx.doi.org/10.3389/fpls.2018.01142
Shi R, Junker A, Seiler C, Altmann T:
Phenotyping roots in darkness: disturbance-free root imaging with near infrared illumination. Funct. Plant Biol. 45 (2018) 400-411. https://doi.org/10.1071/FP17262
scroll top