Meiose
Die unabhängige Arbeitsgruppe Meiose (ME), hauptsächlich vom Europäischen Forschungsrat (Projekt MEIOBARMIX) gefördert, forscht an dem Prozess der pflanzlichen Meiose, der genetische Variation durch homologe Rekombination erzeugt.
Unsere Forschung ist eng mit dem Forschungsinteresse der Abteilung Züchtungsforschung verknüpft und wir tragen mit unseren Arbeiten zum IPK-Forschungsschwerpunkt FSP3 "Reproduktionsmechanismen" bei.
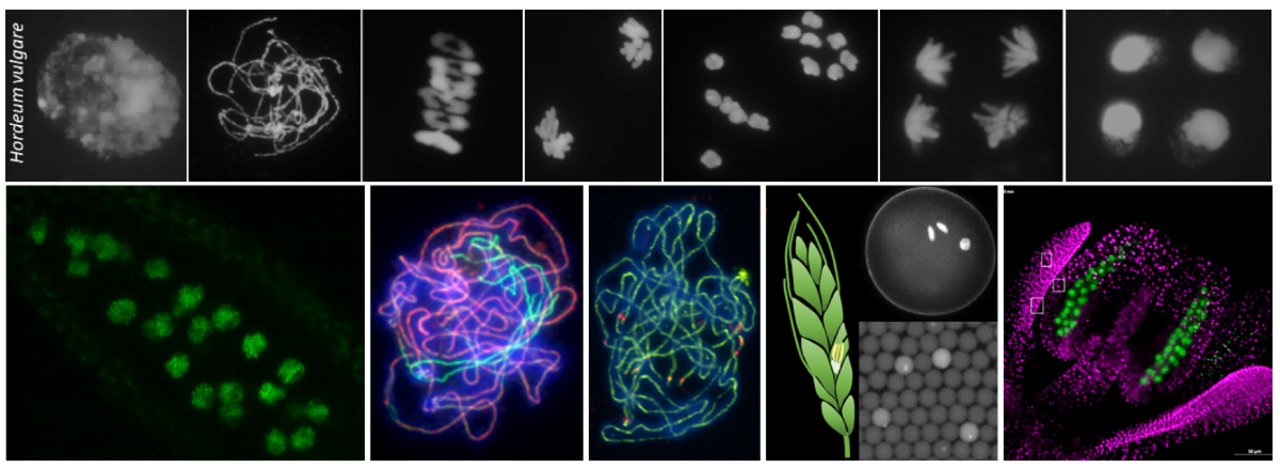
Gerste (Hordeum vulgare) ist eine wichtige Getreidepflanze, die in einer Vielzahl von Klimazonen angebaut und weltweit als Tierfutter, Nahrungsmittel und zum Brauen verwendet wird. Die traditionelle Pflanzenzüchtung, die sich die natürliche genetische Variation, die während der Meiose entsteht, zunutze macht, wird eine Schlüsselrolle bei der Verbesserung von Nutzpflanzen spielen.
Die Meiose erzeugt natürliche genetische Variation durch einen regulierten genetischen Austausch elterlicher Erbinformation basierend auf homologer Rekombination. Allerdings sind insbesondere bei Getreidepflanzen, einschließlich der Gerste, meiotische Rekombinationsereignisse auf die Chromosomenenden beschränkt, wodurch ein Großteil des genetischen Materials in Zuchtprogrammen unangetastet bleibt.
scroll top
Projekte
Aufbauend auf molekulargenetischen, biochemischen und zytogenetischen Ansätzen ist das übergeordnete Ziel der Gruppe das Ergebnis der meiotischen Rekombination insbesondere in der Gerste zu beeinflussen. Um dieses zu erreichen, zielen die Projekte in der Gruppe darauf ab:
- die zugrunde liegenden Mechanismen meiotischer Rekombination in der Gerste und Arabidopsis thaliana besser zu verstehen,
- neue Werkzeuge und Strategien zu entwickeln, um meiotische Rekombination in der Gerste und Arabidopsis thaliana zu untersuchen und zu verändern,
- und erworbenes Wissen in der Gerste anzuwenden.
scroll top
Mitarbeitende
scroll top
Publikationen
Tamilselvan-Nattar-Amutha S, Fuchs J, Heckmann S:
Crystal Digital PCR-based single nucleus pollen genotyping in barley (Hordeum vulgare L.) to measure meiotic crossover rates in defined chromosomal intervals. In: Pavlovic G (Ed.): Digital PCR. Methods and protocols. (Series: Methods in molecular biology, Vol. 2969) New York: Humana New York (2026) 978-1-0716-4766-0, 87-100. https://dx.doi.org/10.1007/978-1-0716-4767-7_6
Cuacos M, Heckmann S:
Diversity favoured: heterozygosity attracts crossovers. Nat. Plants 11 (2025) 1706-1707. https://dx.doi.org/10.1038/s41477-025-02066-x
Feng C, Lorenz J, Dreissig S, Schubert V, Wang B, Hartmann F, Cuacos M, Fernández-Jiménez N, Zhao Z, Eggeling C, Câmara A S, Himmelbach A, Heckmann S:
The synaptonemal complex central element SCEP3 interlinks synapsis initiation and crossover formation in Arabidopsis thaliana. Nat. Plants 11 (2025) 1353-1366. https://dx.doi.org/10.1038/s41477-025-02030-9
Feng J-W, Pidon H, Cuacos M, Lux T, Himmelbach A, Haghi R, Fuchs J, Haberer G, Kuo Y-T, Guo Y, Jayakodi M, Toegelová H, Harpke D, Knauft M, Fiebig A, Maruschewski M, Ronen M, Sharon A, Šimková H, Mayer K F X, Spannagl M, Kumlehn J, Heckmann S, Houben A, Blattner F R, Stein N, Mascher M:
A haplotype-resolved pangenome of the barley wild relative Hordeum bulbosum. Nature 645 (2025) 429-438. https://dx.doi.org/10.1038/s41586-025-09270-x
Schüler D, Lange M, Altmann T, Cuacos M, Arend D, D’Auria J C, Fiebig A, Kumlehn J, Neumann K, Melzer M, Rey-Mazón E, Rolletschek H, Scholz U, Willner E, Reif J C:
Data management in balance – a decade of balancing pragmatism, sustainability and innovation at plant research center IPK Gatersleben. J. Integr. Bioinform. 22 (2025) 20250012. https://dx.doi.org/10.1515/jib-2025-0012
Crhak Khaitova L, Mikulkova P, Pecinkova J, Kalidass M, Heckmann S, Lermontova I, Riha K:
Heat stress impairs centromere structure and segregation of meiotic chromosomes in Arabidopsis. eLife 12 (2024) e90253. https://dx.doi.org/10.7554/eLife.90253
Ahmadli U, Kalidass M, Crhak Khaitova L, Fuchs J, Cuacos M, Demidov D, Zuo S, Pecinkova J, Mascher M, Ingouff M, Heckmann S, Houben A, Riha K, Lermontova I:
High temperature increases centromere-mediated genome elimination frequency and enhances haploid induction in Arabidopsis. Plant Commun. 4 (2023) 100507. https://dx.doi.org/10.1016/j.xplc.2022.100507
Ayoub M A:
Exploration of strategies to alter the meiotic recombination landscape in barley (Hordeum vulgare). (PhD Thesis) Halle/S., Martin-Luther-Universität Halle-Wittenberg, Naturwissenschaftliche Fakultät III Agrar- und Ernährungswissenschaften, Geowissenschaften und Informatik (2023) 107 pp.
Feng C, Roitinger E, Hudecz O, Cuacos M, Lorenz J, Schubert V, Wang B, Wang R, Mechtler K, Heckmann S:
TurboID-based proteomic profiling of meiotic chromosome axes in Arabidopsis thaliana. Nat. Plants 9 (2023) 616-630. https://dx.doi.org/10.1038/s41477-023-01371-7
Piskorz E W, Xu L, Ma Y, Jiang H:
Double-haploid induction generates extensive differential DNA methylation in Arabidopsis. J. Exp. Bot. 74 (2023) 835-847. https://dx.doi.org/10.1093/jxb/erac397
Steckenborn Coria S:
Towards the induction of site-directed meiotic recombination. (PhD Thesis) Halle/S., Martin-Luther-Universität Halle-Wittenberg, Naturwissenschaftliche Fakultät III Agrar- und Ernährungswissenschaften, Geowissenschaften und Informatik (2023) 106 pp.
Steckenborn S, Cuacos M, Ayoub M A, Feng C, Schubert V, Hoffie I, Hensel G, Kumlehn J, Heckmann S:
The meiotic topoisomerase VI B subunit (MTOPVIB) is essential for meiotic DNA double-strand break formation in barley (Hordeum vulgare L.). Plant Reprod. 36 (2023) 1-15. https://dx.doi.org/10.1007/s00497-022-00444-5
Tamilselvan-Nattar-Amutha S, Hiekel S, Hartmann F, Lorenz J, Dabhi R V, Dreissig S, Hensel G, Kumlehn J, Heckmann S:
Barley stripe mosaic virus-mediated somatic and heritable gene editing in barley (Hordeum vulgare L.). Front. Plant Sci. 14 (2023) 1201446. https://dx.doi.org/10.3389/fpls.2023.1201446
Ahn Y-J:
Development and application of Crystal Digital PCR-based single pollen nucleus genotyping to measure meiotic recombination rates in barley (Hordeum vulgare) in high-throughput. (PhD Thesis) Halle/S., Martin-Luther-Universität Halle-Wittenberg, Institut für Agrar- und Ernährungswissenschaften der Naturwissenschaftlichen Fakultät III (2022) 118 pp.
Randall R S, Jourdain C, Nowicka A, Kaduchová K, Kubová M, Ayoub M A, Schubert V, Tatout C, Colas I, Kalyanikrishna, Desset S, Mermet S, Boulaflous-Stevens A, Kubalová I, Mandáková T, Heckmann S, Lysak M A, Panatta M, Santoro R, Schubert D, Pecinka A, Routh D, Baroux C:
Image analysis workflows to reveal the spatial organization of cell nuclei and chromosomes. Nucleus 13 (2022) 277-299. https://dx.doi.org/10.1080/19491034.2022.2144013
Schreiber M, Gao Y, Koch N, Fuchs J, Heckmann S, Himmelbach A, Börner A, Özkan H, Maurer A, Stein N, Mascher M, Dreissig S:
Recombination landscape divergence between populations is marked by larger low-recombining regions in domesticated rye. Mol. Biol. Evol. 39 (2022) msac131. https://dx.doi.org/10.1093/molbev/msac131
Ahn Y-J, Fuchs J, Houben A, Heckmann S:
High throughput measuring of meiotic recombination rates in barley pollen nuclei using Crystal Digital PCR TM. Plant J. 107 (2021) 649-661. https://dx.doi.org/10.1111/tpj.15305
Cuacos M, Heckmann S:
Arabidopsis thaliana. Brassicaceae (Calender Page). In: Houben A (Ed.): Chromosome Biology 2021. 4th Edition. Gatersleben, Germany: IPK (2021) 1.
Cuacos M, Lambing C, Pachon-Penalba M, Osman K, Armstrong S J, Henderson I R, Sanchez-Moran E, Franklin F C H, Heckmann S:
Meiotic chromosome axis remodelling is critical for meiotic recombination in Brassica rapa. J. Exp. Bot. 72 (2021) 3012-3027. https://dx.doi.org/10.1093/jxb/erab035
Ahn Y-J, Cuacos M, Ayoub M A, Kappermann J, Houben A, Heckmann S:
In planta delivery of chemical compounds into barley meiocytes – EdU as compound example. In: Pradillo M, Heckmann S (Eds.): Plant Meiosis: methods and protocols. (Series: Methods in molecular biology, Vol. 2061) New York, NY: Humana Press (2020) 978-1-4939-9817-3, 381-402. https://dx.doi.org/10.1007/978-1-4939-9818-0_27
Ayoub M A, Heckmann S:
Hordeum vulgare. Poaceae (Calender Page). In: Houben A (Ed.): Plant Cytogenetics 2020. 3th Edition. Gatersleben, Germany: IPK (2020) 1.
Chen E C H, Mathieu S, Hoffrichter A, Ropars J, Dreissig S, Fuchs J, Brachmann A, Corradi N:
More filtering on SNP calling does not remove evidence of inter-nucleus recombination in dikaryotic arbuscular mycorrhizal fungi. Front. Plant Sci. 11 (2020) 912. https://dx.doi.org/10.3389/fpls.2020.00912
Desjardins S D, Ogle D E, Ayoub M A, Heckmann S, Henderson I R, Edwards K J, Higgins J D:
MutS homologue 4 and MutS homologue 5 maintain the obligate crossover in wheat despite stepwise gene loss following polyploidization. Plant Physiol. 183 (2020) 1545-1558. https://dx.doi.org/10.1104/pp.20.00534
Dreissig S, Fuchs J, Himmelbach A, Mascher M, Houben A:
Quantification of recombination rate and segregation distortion by genotyping and sequencing of single pollen nuclei. In: Pradillo M, Heckmann S (Eds.): Plant Meiosis: methods and protocols. (Series: Methods in molecular biology, Vol. 2061) New York, NY: Humana Press (2020) 978-1-4939-9817-3, 281-300. https://dx.doi.org/10.1007/978-1-4939-9818-0_20
Dubey R:
Targeted meiotic DNA Double strand break induction using the CRISPR/Cas9 system analyzed via single pollen genotyping. (Master Thesis) Zaragoza, Catalonia, Spain, Spain University of Lleida, Instituto Agronómico Mediterráneo de Zaragoza (IAMZ-CIHEAM) (2020) 70 pp.
Garavello M, Cuenca J, Dreissig S, Fuchs J, Navarro L, Houben A, Aleza P:
Analysis of crossover events and allele segregation distortion in interspecific citrus hybrids by single pollen genotyping. Front. Plant Sci. 11 (2020) 615. https://dx.doi.org/10.3389/fpls.2020.00615
Khosravi S, Dreissig S, Schindele P, Wolter F, Rutten T, Puchta H, Houben A:
Live Cell CRISPR imaging in plant cells with a telomere-specific guide RNA. In: Heinlein M (Ed.): RNA Tagging: methods and protocols. (Series: Methods in molecular biology, Vol. 2166) New York, NY: Humana Press (2020) 1940-6029 (Electronic)1064-3745 (Linking), 343-356. https://dx.doi.org/10.1007/978-1-0716-0712-1_20
Khosravi S, Ishii T, Dreissig S, Houben A:
Application and prospects of CRISPR/Cas9-based methods to trace defined genomic sequences in living and fixed plant cells. Chromosome Res. 28 (2020) 7-17. https://dx.doi.org/10.1007/s10577-019-09622-0
Le Goff S, Keçeli B N, Jeřábková H, Heckmann S, Rutten T, Cotterell S, Schubert V, Roitinger E, Mechtler K, Franklin F C H, Tatout C, Houben A, Geelen D, Probst A V, Lermontova I:
The H3 histone chaperone NASPSIM3 escorts CenH3 in Arabidopsis. Plant J. 101 (2020) 71-86. https://dx.doi.org/10.1111/tpj.14518
Pradillo M, Heckmann S (Eds.):
Plant Meiosis: methods and protocols. (Series: Methods in molecular biology, Vol. 2061) New York, NY: Humana Press (2020) 978-1-4939-9817-3, 410 pp. https://dx.doi.org/10.1007/978-1-4939-9818-0
Schmidt C, Fransz P, Rönspies M, Dreissig S, Fuchs J, Heckmann S, Houben A, Puchta H:
Changing local recombination patterns in plants by CRISPR/Cas mediated chromosome engineering. Nat. Commun. 11 (2020) 4418. https://dx.doi.org/10.1038/s41467-020-18277-z
Schubert V, Neumann P, Marques A, Heckmann S, Macas J, Pedrosa-Harand A, Schubert I, Jang T-S, Houben A:
Super-resolution microscopy reveals diversity of plant centromere architecture. Int. J. Mol. Sci. 21 (2020) 3488. https://dx.doi.org/10.3390/ijms21103488
Valuchova S, Mikulkova P, Pecinkova J, Klimova J, Krumnikl M, Bainar P, Heckmann S, Tomancak P, Riha K:
Imaging plant germline differentiation within Arabidopsis flowers by light sheet microscopy. eLife 9 (2020) e52546. https://dx.doi.org/10.7554/eLife.52546
Demidov D, Heckmann S, Weiss O, Rutten T, Tomaštíková E D, Kuhlmann M, Scholl P, Municio C M, Lermontova I, Houben A:
Deregulated phosphorylation of CENH3 at Ser65 affects the development of floral meristems in Arabidopsis thaliana. Front. Plant Sci. 10 (2019) 928. https://dx.doi.org/10.3389/Fpls.2019.00928
Dreissig S, Mascher M, Heckmann S:
Variation in recombination rate is shaped by domestication and environmental conditions in barley. Mol. Biol. Evol. 36 (2019) 2029–2039. https://dx.doi.org/10.1093/molbev/msz141
Ishii T, Schubert V, Khosravi S, Dreissig S, Metje-Sprink J, Sprink T, Fuchs J, Meister A, Houben A:
RNA-guided endonuclease – in situ labelling (RGEN-ISL): a fast CRISPR/Cas9-based method to label genomic sequences in various species. New Phytol. 222 (2019) 1652-1661. https://dx.doi.org/10.1111/nph.15720
Chen E C, Mathieu S, Hoffrichter A, Sedzielewska-Toro K, Peart M, Pelin A, Ndikumana S, Ropars J, Dreissig S, Fuchs J, Brachmann A, Corradi N:
Single nucleus sequencing reveals evidence of inter-nucleus recombination in arbuscular mycorrhizal fungi. eLife 7 (2018) e39813. https://dx.doi.org/10.7554/eLife.39813
Lambing C, Heckmann S:
Tackling plant meiosis: from model research to crop improvement. Front. Plant Sci. 9 (2018) 829. https://dx.doi.org/10.3389/fpls.2018.00829
Kappermann J:
Beeinflussung der Anzahl und Verteilung meiotischer Rekombinationsereignisse in der Gerste (Hordeum vulgare) mittels Manipulation epigenetischer Faktoren anhand von Chemikalien. (Bachelor Thesis) Köthen, Hochschule Anhalt (2017)
scroll top