Forschungsgebiete
Leitung: Dr. habil. Helmut Bäumlein
Im Mittelpunkt unseres Interesses steht die molekulare Analyse genetischer und epigenetischer Prozesse, die der sexuellen und asexuellen Reproduktion zu Grunde liegen. Dazu arbeiten wir an der sexuellen Modellpflanze Arabidopsis, an parthenogenetischen Linien des Weizen `Salmon` Systems sowie an den Apomikten Hypericum und Boechera.
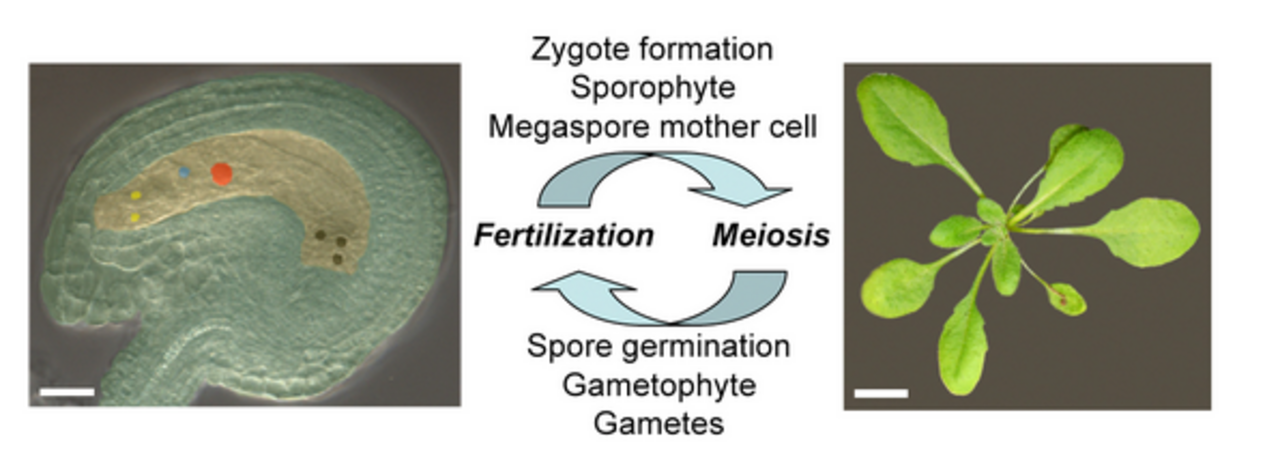
Pflanzliche Entwicklung ist durch die alternierende Folge zweier Generationen gekennzeichnet, dem Gameten-bildenden Gametophyten und dem Sporen-bildenden Sporophyten. Meiotische Reduktion diploider Sporen führt zur Bildung weiblicher und männlicher Gameten im weiblichen (Embryosack) bzw. männlichen (Pollen) Gametophyten. Bei der Befruchtung fusionieren weibliche und männliche Gameten (Eizelle bzw. Spermazelle) zur Zygote, aus der ein diploider sporophytischer Embryo entsteht. Charakteristisches Merkmal der Blütenpflanzen ist die doppelte Befruchtung. Dabei wird ein zweiter weiblicher Gamet, die homo-diploide Zentralzelle, durch eine weitere Spermazelle befruchtet. Das triploide Befruchtungsprodukt entwickelt sich zum Endosperm, ein der Ernährung des Embryos dienendes Nährgewebe. Während der anschließenden Samenreifung werden Speicherstoffe deponiert; es kommt zur Ausprägung von Trocknungstoleranz und Dormanz. Aus dem nach einer Ruhephase keimenden Samen entsteht der bei höheren Pflanzen bezüglich der Biomasse stark dominierende neue Sporophyt.
Die beschriebene sexuelle Reproduktion ist charakteristisch für die meisten Pflanzen. Einige Pflanzenarten vermehren sich jedoch durch asexuelle Samenbildung, ein Prozess, der als Apomixis bezeichnet wird. Durch Apomeiose und Parthenogenese werden dabei Meiose bzw. Befruchtung vermieden bzw. umgangen, was zu genetischen Klonen der Mutterpflanze führt. Die molekulare Analyse dieser sexuellen bzw. asexuellen Entwicklungsprozesse ist einerseits ein faszinierender Aspekt grundlegender Entwicklungsbiologie, andererseits ein seit Langem angestrebtes Problem der biotechnologischen Anwendung, insbesondere für die Produktion von Nahrungsmitteln sowie die Fixierung von Hybrideffekten (d.h. Heterosis) durch Apomixis.